EPT-Net:用于3D医学图像分割的边缘感知转换器
IEEE TRANSACTIONS ON MEDICAL IMAGING, VOL. 42, NO. 11, NOVEMBER 2023
背景
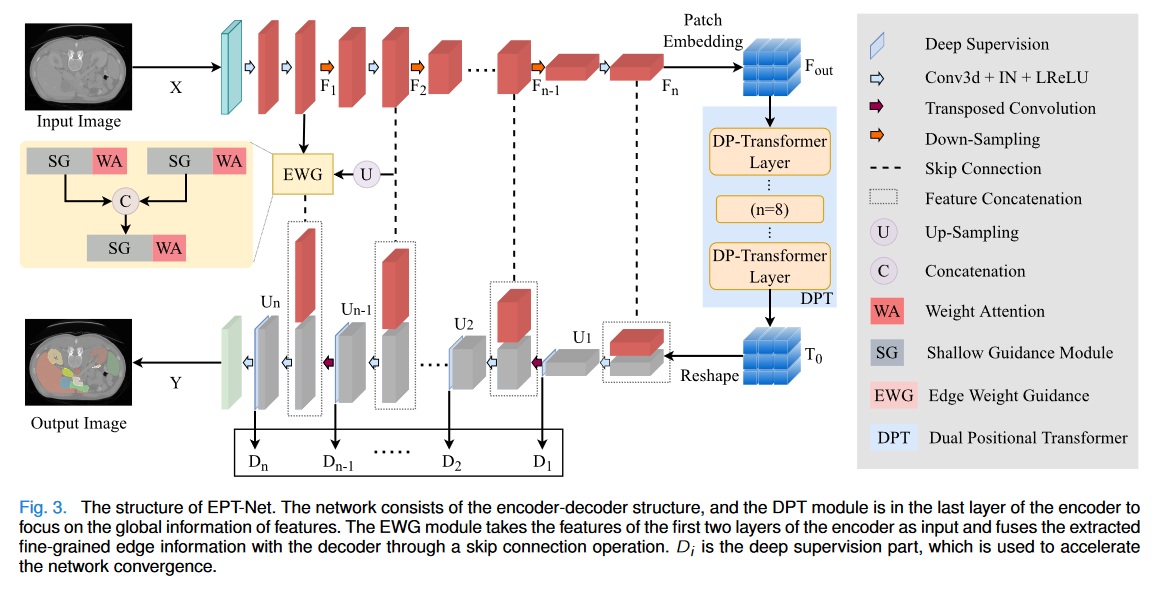
卷积运算的内在局部性在建模长程依赖性方面存在局限性。尽管为序列到序列全局预测而设计的Transformer就是为了解决这个问题而诞生的,但由于底层细节特征不足,它可能会导致定位能力有限。此外,低级特征具有丰富的细粒度信息,这极大地影响了不同器官的边缘分割决策。然而,一个简单的CNN模块很难捕捉细粒度特征中的边缘信息,并且在处理高分辨率3D特征时消耗的计算能力和内存是昂贵的。
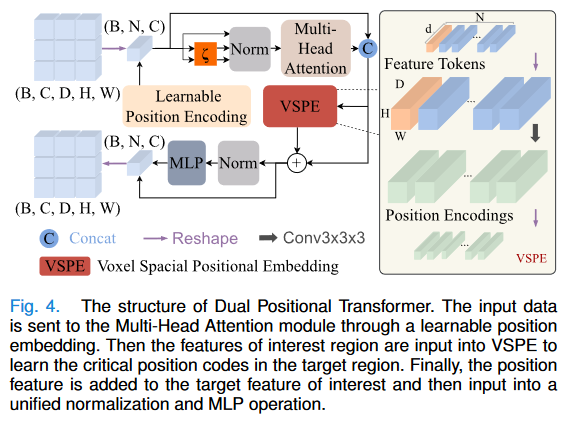
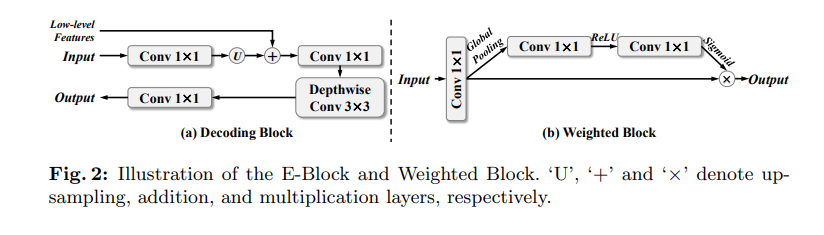
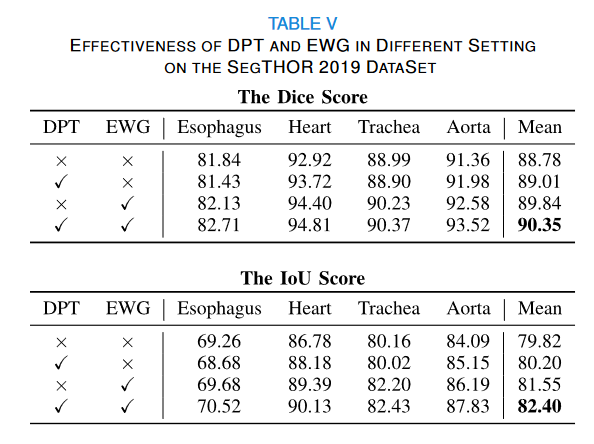
本文提出了一种双位置变换器,以有效地提高三维空间定位能力。此外,由于低级别特征包含详细信息,我们在不添加网络参数的情况下,通过最小化边缘信息函数来执行边缘权重引导模块来提取边缘信息
贡献
实验
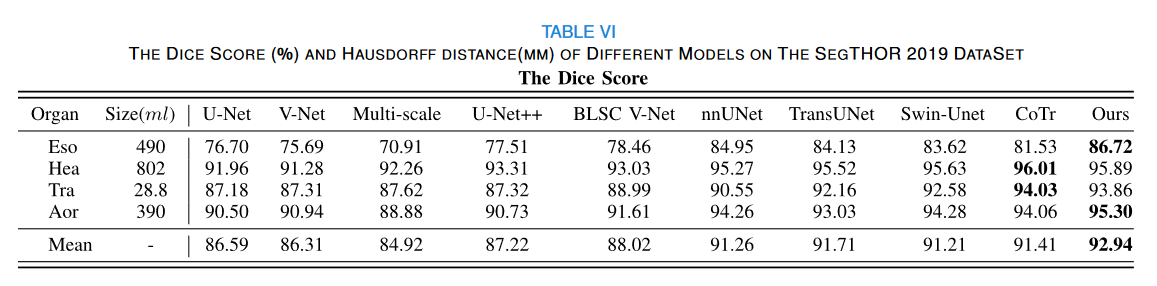
选择了三个数据集进行实验验证,其中包括两个公共数据集SegTHOR 2019[17],颅骨(BCV)之外的多图谱标记[18]和重新标记的KiTS19数据集[19],称为KiTS19-M。首先,介绍了SegTHOR 2019数据集,并专门用于在放疗期间分割肿瘤周围胸腔中的危险器官,包括心脏(hea)、气管(tra)、主动脉(aor)和食道(eso)。该数据集包括60个CT扫描,分为40个训练集和20个测试集。经验丰富的放射治疗师已经手动绘制了心脏、气管、主动脉和食道。接下来,MultiAtlas Labeling Beyond the Cranial Vault包含13个腹部器官的标签,包括脾脏(spl)、左右肾(L-K和R-K)、胆囊(gal)、食道(eso)、肝脏(liv)和胃(sto)、主动脉(aor)、下腔静脉(I-V-C)、门静脉和脾静脉(P-V/S-V)、胰腺(pan)、左右肾上腺(R-A-G和L-A-G),由两名经验丰富的大学生和放射科医生手工标记。使用MIPAV软件进行容量验证。该数据集包含30个标记的CT图像和20个测试图像。KiTS19-M数据集来自KiTS19数据集肾脏肿瘤分割竞赛。它包含300个样本,包括210个训练样本和90个测试样本。为了促进我们对多器官分割的研究,我们对数据集中的多个器官进行了重新标记。经过经验丰富的专家重新标记,我们对四个腹部器官进行了精细标记,包括肝、脾、右肾和左肾。
方法
U型网络结构在医学图像分割中取得了显著的成就。由于卷积运算的内在局部性和编码器-解码器的U型网络结构中缺乏全局上下文边缘信息处理,我们提出了EPT-Net。该网络主要基于U型网络,由双位置Transformer(DPT)和边缘权重引导(EWG)模块组成。DPT是在编码器末端提取深度信息,通过Transformer解决了U型网络的远程依赖性限制。将浅层细粒度特征输入EWG以提取图像边缘信息。