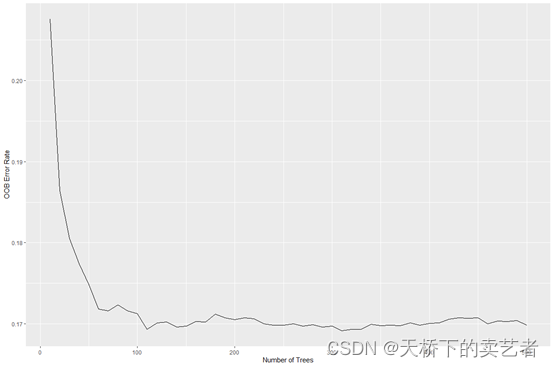
随机森林(Breiman 2001a)(RF)是一种非参数统计方法,需要没有关于响应的协变关系的分布假设。RF是一种强大的、非线性的技术,通过拟合一组树来稳定预测精度模型估计。随机生存森林(RSF)(Ishwaran和Kogalur,2007;Ishwaraan,Kogalur、Blackstone和Lauer(2008)是Breimans射频技术的延伸从而降低了对时间到事件数据的有效非参数分析。
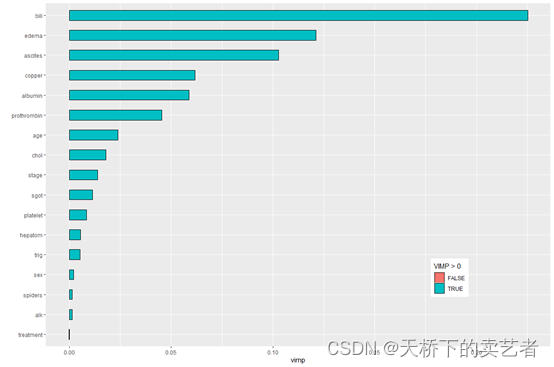
R语言随机森林进行生存分析需要使用到randomForestSRC包,是对Breimans随机森林的统一处理用于生存、回归和分类问题。randomForestSRC包还有一个用于做图的ggRandomForests包,搭配使用,今天咱们来介绍一下怎么使用randomForestSRC包进行随机森林生存分析,内容有点多,咱们分2章来介绍。
咱们先导入数据和R包
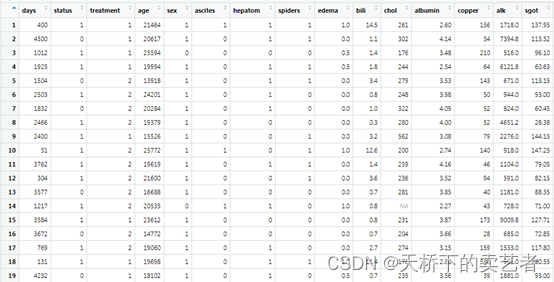
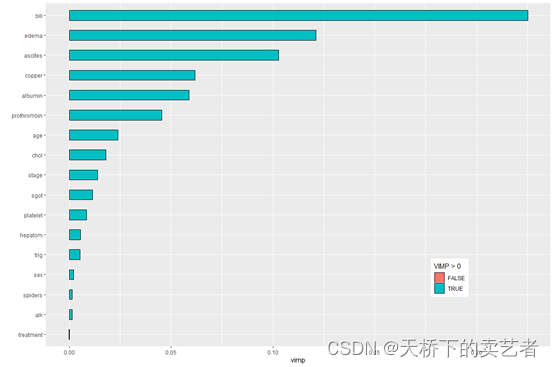
这是一个胆管炎数据(公众号回复:胆管炎数据2,可以获得数据),years:生存时间,status:结局指标,是否死亡,treatment是否DPCA治疗,age年龄,sex性别,ascites是否有腹水,hepatom是否有肝肿大,spiders是否有蜘蛛痣,edema水肿的级别,bili胆红素,chol胆固醇,albumin白蛋白,copper尿酮,alk碱性磷酸酶,sgot:SGOT评分,trig甘油三酯,platelet血小板,prothrombin凝血酶时间,stage组织学分型
我们对数据处理一下,把treatment这个变量变成因子
接下来咱们把数据分成两组,有treatment数据的为测试组,treatment数据缺失的为对照组。
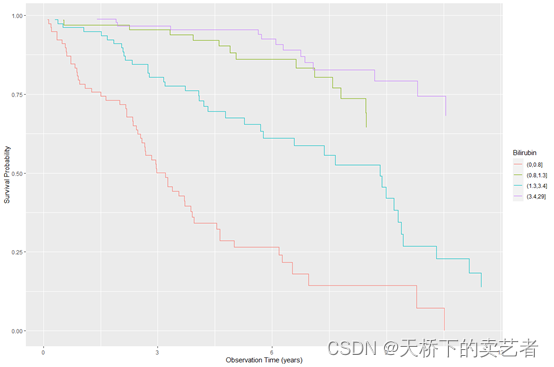
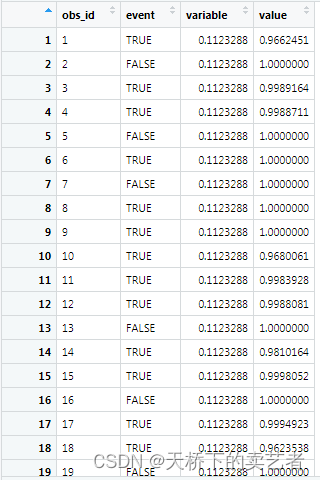
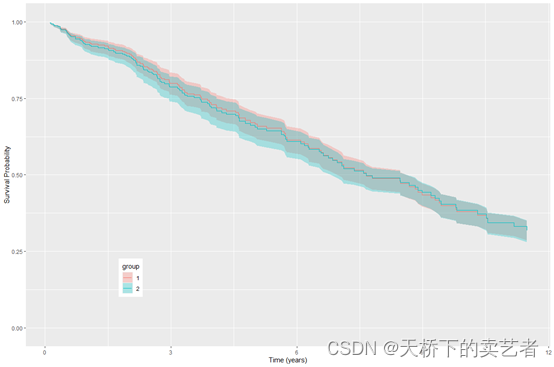
我们先用生存分析做一遍,等下可以和随机森林进行比较,接下来我们用gg_survival对测试组生成生存分析的数据,这个函数挺方便使用的,生成了生存分析的详尽数据
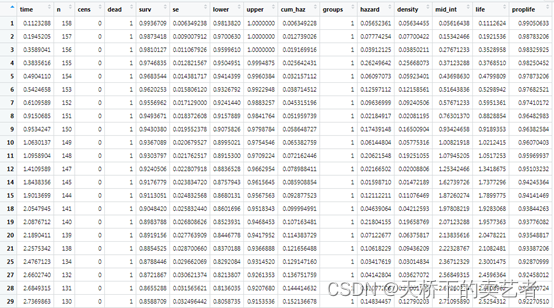
绘图
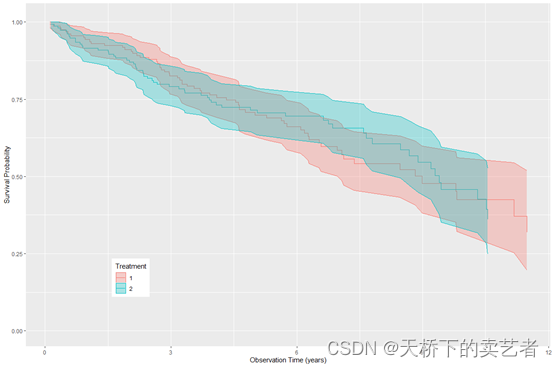
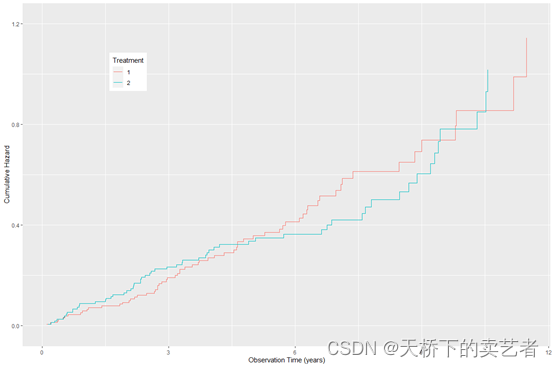
或者绘制成这种累积风险图